珠海舒桐医疗科技有限公司
5 年
手机商铺
- NaN
- 0.5999999999999996
- 0.5999999999999996
- 1.5999999999999996
- 0.5999999999999996
珠海舒桐医疗科技有限公司
入驻年限:5 年
- 联系人:
柳嘉欣
- 所在地区:
广东 珠海市 香洲区
- 业务范围:
试剂、技术服务、抗体、细胞库 / 细胞培养、ELISA 试剂盒、耗材、原辅料包材
- 经营模式:
科研机构 生产厂商
推荐产品
公司新闻/正文
Molecular Plant Pathology重磅:基于Tn5转座酶的ATAC-seq联合RNA-seq,系统揭示水稻应对条纹病毒侵染的核心机制
138 人阅读发布时间:2025-10-20 14:48
一、技术背景与研究意义
随着高通量测序技术的快速发展,表观基因组学与转录组学的整合分析已成为解析生物过程调控机制的重要手段。ATAC-seq(Assay for Transposase-Accessible Chromatin using sequencing)是一种用于全基因组范围内检测染色质开放性的技术,它通过Tn5转座酶对开放染色质区域进行特异性切割并引入测序接头,从而实现对染色质可及性的高分辨率定位。RNA-seq(RNA sequencing)则通过对转录本进行测序,全面揭示基因表达水平的变化。将ATAC-seq与RNA-seq数据整合分析,能够从染色质开放状态与基因表达水平两个维度,系统阐释基因调控网络,尤其在植物—病原互作研究中具有重要价值。
水稻作为全球最重要的粮食作物之一,其生产受到多种病毒病的威胁。水稻条纹病毒(Rice Stripe Virus, RSV)是由灰飞虱传播的恶性病毒,可引起水稻叶片黄化条纹、植株矮化等症状,导致严重产量损失。然而,目前关于RSV感染如何影响植物染色质结构及其与宿主基因表达调控的关系,尚缺乏系统研究。
本研究首次将ATAC-seq与RNA-seq技术联合应用于RSV感染的水稻模型中,系统分析了病毒侵染引起的染色质可及性变化及其对基因表达的影响,并进一步筛选出与RSV蛋白互作的关键转录因子,为解析植物—病毒互作的分子机制提供了新视角。相关研究发表在期刊《Molecular Plant Pathology》上,题目为《Integrated ATAC-seq and RNA-seq data analysis identifies transcription factors related to rice stripe virus infection in Oryza sativa》。

二、整合分析技术的核心优势
ATAC-seq与RNA-seq的整合分析具有以下几大核心优势:
-
多层次数据互补:ATAC-seq揭示染色质开放性变化,RNA-seq反映基因表达水平,两者结合可识别出受染色质状态调控的差异表达基因,提高候选基因筛选的准确性。
-
高分辨率与全基因组覆盖:ATAC-seq可在全基因组范围内识别开放染色质区域,包括启动子、增强子等调控元件,结合RNA-seq可精准定位功能性调控事件。
-
无需先验知识:该技术不依赖于已知的调控信息,适用于探索性研究,特别适合在植物—病原互作等复杂系统中发现新的调控因子。
-
支持下游功能验证:通过整合分析筛选出的候选基因或转录因子,可直接用于后续的蛋白互作、基因功能验证等实验,形成完整的研究闭环。
本研究正是利用上述优势,成功识别出多个在RSV感染中起关键作用的转录因子,并验证了其与病毒蛋白的相互作用。
三、关键实验结果与方法解析
1. RSV感染下水稻染色质可及性全景图谱
本研究通过ATAC-seq技术对RSV感染与未感染(Mock)水稻幼苗的染色质开放性进行检测。样本经Tn5转座酶处理后构建文库,进行Illumina高通量测序。使用MACS2软件进行peak calling,识别出可及染色质区域(Accessible Chromatin Regions, ACRs),并通过Circos软件展示其在染色体上的分布情况。此外,对转录起始位点(Transcription Start Sites, TSS)及其上下游3 kb区域的ATAC-seq信号进行了分析。
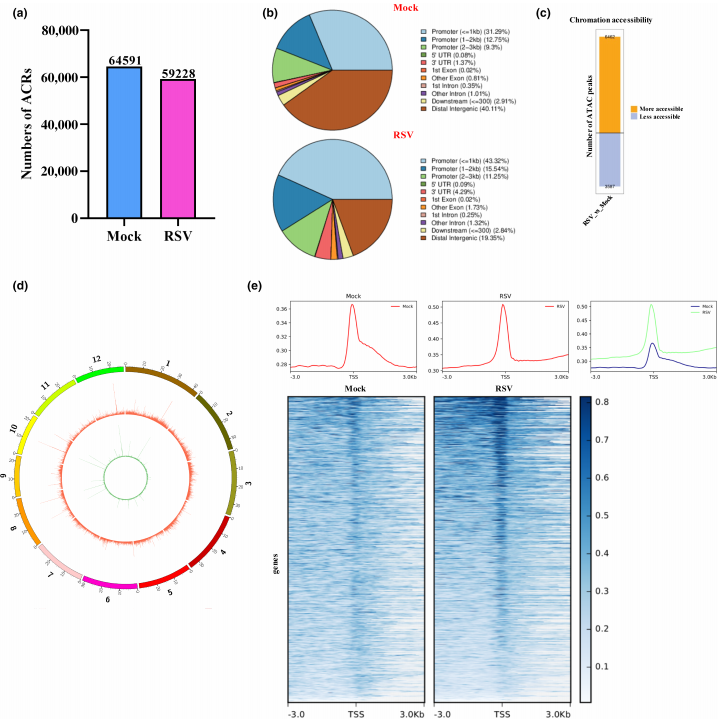
图1 水稻条纹叶枯病毒(RSV)感染下水稻幼苗的染色质可及性景观
重要实验结论:
-
RSV感染导致染色质开放性发生显著变化,共识别出59,228个ACRs(感染组)与64,591个ACRs(Mock组),其中6462个区域开放性增加,3587个区域开放性降低(图1a, 1c)。
-
ACRs主要富集在启动子区和远端 intergenic 区域,提示这些区域在转录调控中具有重要作用(图1b)。
-
TSS区域显示出最高的染色质可及性信号,表明该区域在基因表达调控中具有核心地位(图1e)。
2. RSV感染相关ACR基因的功能富集分析
为探究在RSV感染中发生开放性变化的ACR所关联基因的生物学功能,研究团队对开放与关闭ACR相关基因分别进行了GO(Gene Ontology)与KEGG(Kyoto Encyclopedia of Genes and Genomes)通路富集分析。GO分析涵盖生物过程、分子功能与细胞组分三个类别,KEGG分析则聚焦于代谢与信号通路。
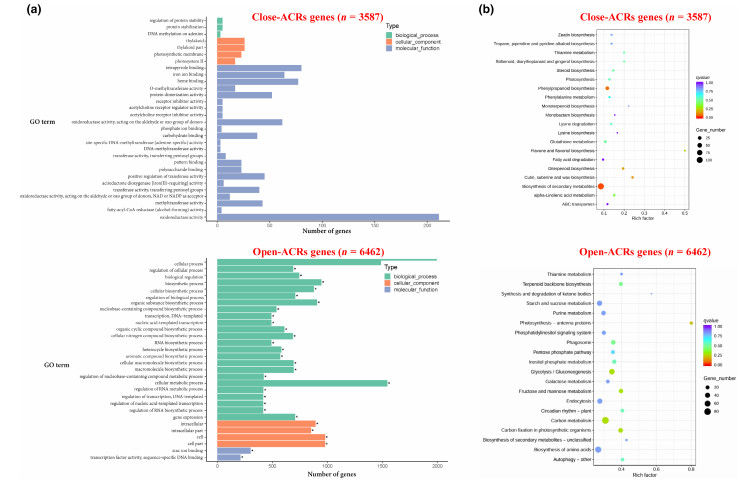
图2 水稻条纹叶枯病毒(RSV)感染下可及染色质区域(ACR)相关基因的分析
重要实验结论:
-
开放ACR相关基因显著富集于细胞代谢过程、生物调控等生物过程,以及碳代谢、氨基酸生物合成等通路(图2a, 2b)。
-
关闭ACR相关基因在氧化还原酶活性、次级代谢产物合成等生物合成通路中显著富集,暗示RSV感染可能影响植物的抗氧化与次生代谢响应(图2a, 2b)。
3. ATAC-seq与RNA-seq整合分析识别关键基因
本研究进一步对Mock与RSV感染的水稻样本进行RNA-seq分析,使用DESeq2筛选差异表达基因(DEGs)。将ATAC-seq与RNA-seq数据整合后,筛选出在开放DACR(Differentially Accessible Chromatin Regions)中表达上调、在关闭DACR中表达下调的基因,共获得349个上调基因与126个下调基因。
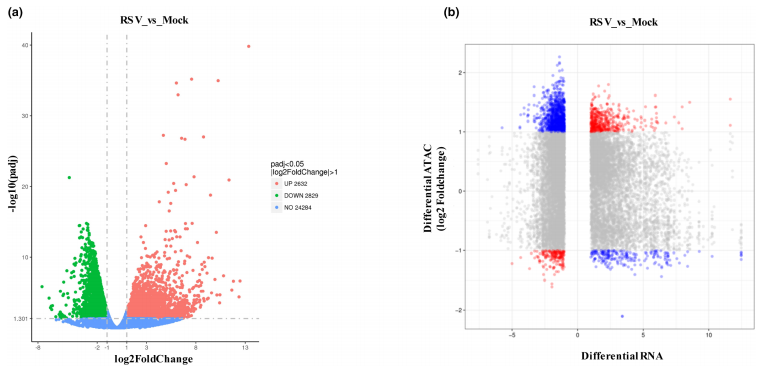
图3 ATAC-seq与RNA-seq的整合分析
重要实验结论:
-
共识别出5461个DEGs,其中2632个基因上调,2829个基因下调(图3a)。
-
整合分析共筛选出475个受染色质状态变化直接调控的差异表达基因,其中包括349个在开放区域中上调的基因和126个在关闭区域中下调的基因,这些基因可能在RSV感染或宿主—病毒互作中发挥关键作用(图3b)。
4. DEGs的功能与通路富集分析
对整合分析筛选出的DEGs进行GO与KEGG富集分析,以揭示其在RSV感染中的生物学功能与参与的主要代谢与信号通路。
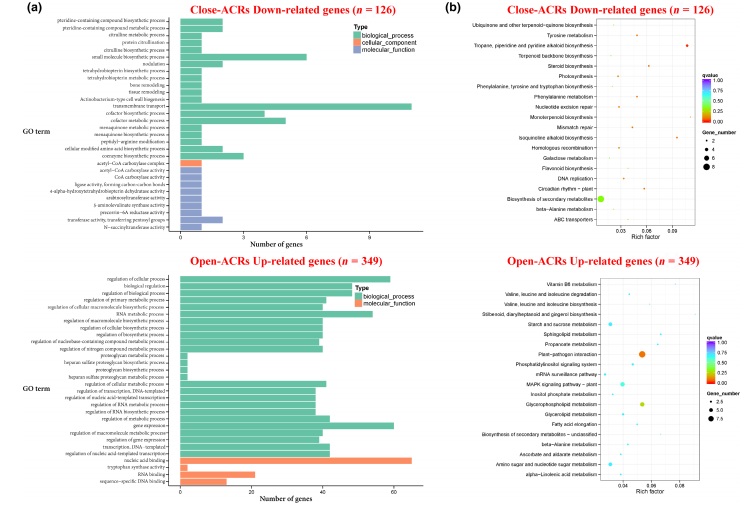
图4 可及染色质区域(ACRs)中差异表达基因(DEGs)的GO和KEGG通路富集分析
重要实验结论:
-
GO分析显示,DEGs显著富集于瓜氨酸生物合成、细胞壁形成、核酸结合、生物调控等过程,(图4a),结果表明细胞壁组分和某些重要氨基酸的生物合成可能为响应RSV感染而启动。
-
KEGG分析表明,植物—病原互作、甘油磷脂代谢、MAPK信号通路、淀粉与蔗糖代谢等通路在RSV感染中显著激活,这些代谢过程可能在水稻响应RSV感染中发挥重要作用(图4b)。
5. 转录因子的验证与足迹分析
研究团队通过搜索ATAC-seq数据中的上游 motif,识别出34个可能与RSV感染相关的转录因子(Transcription Factors, TFs)。从中选取8个已报道与植物抗逆相关的TF(如OsWRKY77、OsERF91等),利用RT-qPCR验证其表达水平。同时,使用TOBIAS软件对ATAC-seq数据进行转录因子足迹分析,预测TF结合位点的占用状态。
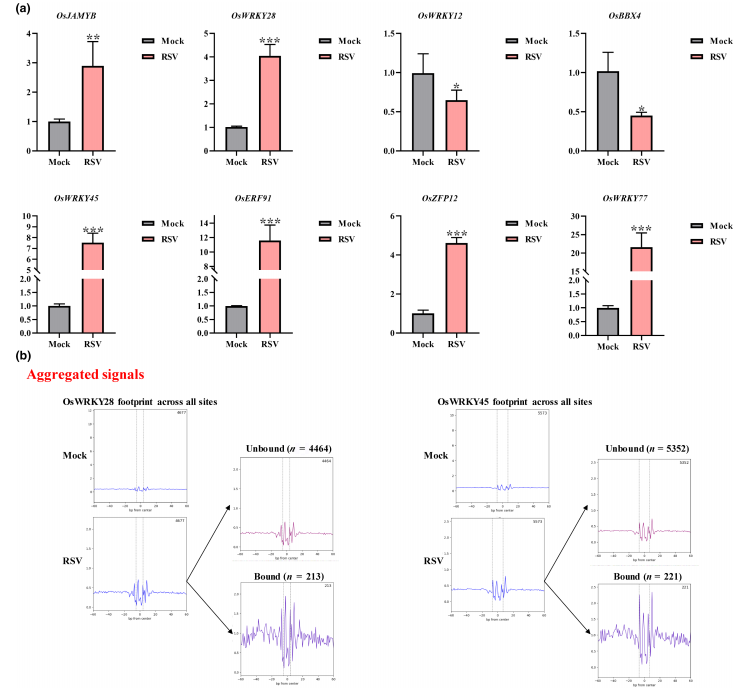
图5 分别使用逆转录定量PCR(RT-qPCR)和TOBIAS对基于ATAC-seq和RNA-seq鉴定的转录因子(TFs)进行验证和可视化
重要实验结论:
-
RT-qPCR结果与RNA-seq一致,RSV感染后OsWRKY12与OsBBX4表达下调,其余6个TF表达上调(图5a)。
-
TOBIAS足迹分析显示,RSV感染后OsWRKY28与OsWRKY45在RSV感染后结合位点占用状态发生改变,提示其可能参与病毒诱导的转录重编程(图5b)。
6. 转录因子与RSV蛋白的相互作用验证
为验证筛选出的TF是否直接参与RSV感染过程,研究采用双分子荧光互补(BiFC)、荧光素酶互补成像(LCI)及免疫共沉淀(Co-IP)等技术,系统分析TF与RSV蛋白之间的互作关系。
表1 鉴定的转录因子与水稻条纹叶枯病毒(RSV)蛋白之间的相互作用
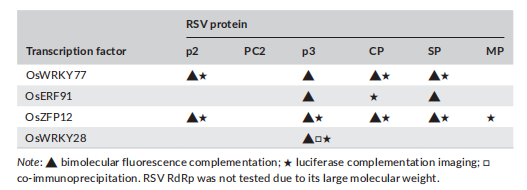
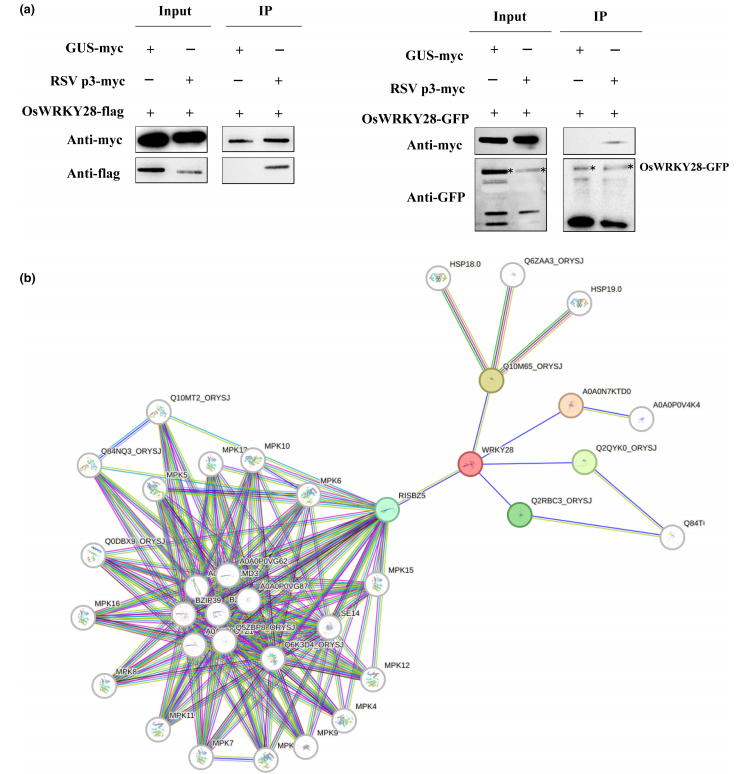
图6 鉴定的转录因子与水稻条纹叶枯病毒(RSV)蛋白之间的相互作用
重要实验结论:
- OsWRKY77、OsERF91、OsZFP12与OsWRKY28均与至少一种RSV蛋白发生相互作用(表1)。
- sWRKY28与RSV p3蛋白的互作关系在Co-IP实验中得到进一步确认(图6a)。
- l 通过STRING数据库预测OsWRKY28与MAPK激酶及bZIP类转录因子存在互作网络,提示其可能位于免疫调控网络的核心位置(图6b)。
四、研究总结与展望
本研究首次将ATAC-seq与RNA-seq技术联合应用于RSV感染的水稻系统中,系统揭示了病毒引起的染色质开放性变化及其对基因表达的调控作用。通过整合分析,不仅识别出大量受染色质状态调控的差异表达基因,还进一步筛选出4个与RSV蛋白直接互作的关键转录因子(OsWRKY77、OsWRKY28、OsZFP12与OsERF91),为理解植物—病毒互作的分子机制提供了新的实验依据。
该研究策略展示了多组学整合分析在植物抗病研究中的强大潜力,尤其适用于识别上游调控因子与信号通路节点。未来,此类方法可扩展至其他作物—病原系统中,为抗病育种与绿色防控提供新靶点。
五、舒桐科技:以自主核心技术,赋能植物抗病多组学研究
作为基因组学研究领域的前沿企业,珠海舒桐医疗科技有限公司一直致力于为科研工作者提供高精度的技术支持和高品质的研究工具,为客户提供从核心酶原料到完整解决方案的一站式多组学服务。
自主核心原料,保障数据源头卓越
我们深知技术根基的重要性。舒桐科技拥有自主知识产权的高性能Tn5转座酶生产工艺,确保其在ATAC-seq等应用中具备高活性、低背景噪音的优异表现。这使我们能够从源头上保证染色质开放性研究数据的可靠性与准确性。
一体化解决方案,打通研究全链条
基于自产的核心酶原料,我们开发了系列高性能建库试剂盒,并可提供涵盖 ATAC-seq、RNA-seq、ChIP-seq、CUT&Tag-seq、WGS在内的全方位组学检测服务。我们具备专业的生物信息学团队,能够为您提供从课题设计、文库构建、高通量测序到深度数据整合分析与机制挖掘的全流程服务,真正实现“样品入,答案出”的无缝对接。
深耕农业科学,专注解决实际问题
我们特别关注植物科学领域的挑战。如果您正从事植物—病原互作、抗逆机制解析、表观遗传调控或关键基因/转录因子功能研究,舒桐科技将依托自身技术平台与项目经验,为您量身定制最优的多组学研究方案,助力您在作物抗病育种与基础研究领域取得关键突破。
如果您正从事植物—病原互作、抗逆机制或表观遗传调控相关研究,欢迎联系舒桐科技,我们将为您定制专属的多组学研究方案,助力您在基因组学领域或作物抗病领域取得突破性成果。